
钍基核裂变能全国重点实验室 |
|||
|
-钍基核能物理中心-
|
|||
熔盐机械工程技术部 |
|||
仪控工程技术部 |
|||
熔盐化学工程技术部 |
|||
|
-核能综合利用研究中心-
|
|||
材料研究部 |
|||
钍铀循环化学部 |
|||
应用化学技术部 |
|||
氚科学与工程技术部 |
|||
核与辐射安全技术部 |
|||
应用加速器技术部 |
|||
反应堆运行技术部 |



2014年10月23日,清华大学医学院基础医学系和结构生物学中心李海涛研究团队在国际学术期刊《细胞》(Cell)发表了题为“AF9 YEATS domain links histone acetylation to DOT1L-mediated H3K79 methylation”(AF9 YEATS结构域关联组蛋白乙酰化至DOT1L介导的组蛋白H3第79位赖氨酸甲基化)的研究论文,发现了一种新型组蛋白乙酰化阅读器-YEATS结构域,揭开了组蛋白乙酰化转录调控研究新篇章。
真核生物基因组DNA 是以染色质形式存在的。染色质凝聚、松弛结构的形成,以及开放、闭合等状态的转换提供了一种超越DNA 序列本身的调控机制,即表观遗传调控,其具体机制涉及组蛋白修饰、组蛋白变体、DNA 甲基化、非编码RNA,及染色质重塑等。其中,组蛋白翻译后修饰被认为构成一类超越基因序列的“组蛋白密码”,控制着遗传信息的组织层次及其在染色质层面的解读。组蛋白赖氨酸乙酰化是研究最早的一类组蛋白修饰,与基因活化关系密切,但很长一段时间内溴域(Bromodomain)是唯一一个为人所知的乙酰化组蛋白阅读器,数个溴域靶向的一类新药已处于临床试验阶段。因此,发现新型乙酰化组蛋白阅读器一直是表观遗传学领域的研究热点。
李海涛研究组本项研究首次证明YEATS结构域是一类新型组蛋白乙酰化修饰“阅读器”。利用上海光源生物大分子晶体学线站(BL17U1),最终得以及时解析了2.3埃分辨率的AF9蛋白YEATS结构域与组蛋白H3K9ac(组蛋白H3赖氨酸9位乙酰化)修饰多肽复合物的晶体结构,阐释了AF9蛋白YEATS结构域特异H3K9ac修饰的分子机制。研究发现,AF9YEATS具有免疫球蛋白家族八链β三明治骨架,与由四股α螺旋构成的溴域截然不同,代表了一种全新折叠类型的组蛋白阅读器。
AF9基因和MLL基因的融合(MLL-AF9)作为最常见的MLL重排类型,是导致人类急性髓细胞白血病(AML)以及急性淋巴性白血病(ALL)等疾病的驱动性因素。进一步的细胞生物学和功能基因组学研究发现,人AF9与H3K9ac修饰在全基因组水平有强烈的共定位,并且调控了包括MYC、BMP2、HOXA基因簇在内的细胞增殖分化基因的表达;此外,本研究首次表明AF9通过识别组蛋白H3K9乙酰化修饰,招募DOTL1到特定染色质区段,促进了组蛋白H3K79甲基化的共沉积和基因活化,揭示出一种新型组蛋白修饰交叉会话机制,体现了真核生物表观遗传调控的复杂和精密性。
目前自然界中鉴定出的含有YEATS结构域的蛋白已达数百个,遍布于70多个不同物种,从酵母到人类都保守存在。含有YEATS结构域的蛋白因子,如AF9、ENL、GAS41等,不仅与转录调控密切相关,它们的调控异常通常会导致白血病、癌症等人类疾病。值得注意的是,虽然YEATS与溴域拥有类似的乙酰化赖氨酸识别功能,目前针对溴域乙酰化识别口袋的抑制剂(+)-JQ1对YEATS毫无效果,体现了YEATS乙酰化识别口袋的独特性。因此,本研究成果不仅揭示了组蛋白乙酰化调控和人类疾病与健康的关系,也为后续YEATS结构域靶向的小分子药物开发奠定了基础。
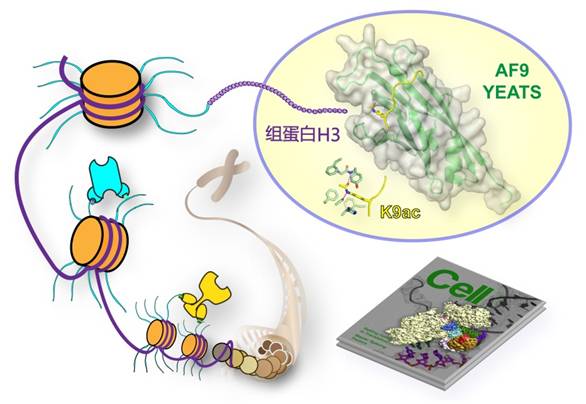
图为AF9 YEATS结构域(绿色)识别H3K9乙酰化多肽(黄色)参与表观遗传转录激活调控。